
DNA-bindende Proteine
Ein DNA-bindendes Protein ist ein Protein, das an DNA bindet.
Inhaltsverzeichnis
Eigenschaften
DNA-bindende Proteine kommen in allen Lebewesen und DNA-Viren vor. Sie besitzen mindestens eine Proteindomäne, welche an DNA binden kann. Die Bindung kann dabei an verschiedenen funktionellen Gruppen erfolgen. Das Rückgrat der DNA besteht aus sich abwechselnden Phosphat- und Desoxyribose-Einheiten. Durch die Phosphatgruppen ist die DNA proportional zur Kettenlänge mit negativen Ladungen versehen, an die unter anderem Proteindomänen mit positiv-geladenen Aminosäuren (z. B. Lysin, Arginin) oder Proteindomänen mit negativ geladenen Aminosäuren mit komplexierten Kationen binden können (z. B. Mg2+- oder Zn2+-Komplexe). Da das Desoxyribosephosphat-Rückgrat sich ständig wiederholt, kann ein ausschließlich an das Rückgrat bindendes Protein nicht an eine bestimmte DNA-Sequenz binden (keine Sequenzspezifität). Eine sequenzspezifische Bindung erfolgt durch zumindest teilweise Bindung an eine bestimmte Folge von Nukleinbasen, teilweise kann auch das Rückgrat gebunden werden.
Sequenzspezifität
DNA-bindende Proteine ohne Sequenzspezifität sind z. B. die Polymerasen, Helicasen und generell Proteine, die an der DNA entlanggleiten (z. B. mit DNA-Klammer). Sequenzspezifisch DNA-bindende Proteine sind z. B. Transkriptionsfaktoren, die an definierten Stellen (Promotor) eine Genexpression auslösen. Aufgrund des höheren Anteils an Nukleinbasen in der Oberfläche binden sequenzspezifische DNA-bindende Moleküle eher in der großen Furche der DNA-Doppelhelix. Während manche sequenzspezifisch DNA-bindende Proteine bevorzugt einzelsträngige DNA binden (z. B. Einzelstrang-bindendes Protein), binden andere doppelsträngige DNA (die meisten) und einige wenige auch Heteroduplexe aus DNA und RNA (z. B. Telomerase, Reverse Transkriptasen, RNase H).
Bindung einzelsträngiger DNA
Einzelsträngige DNA kommt in Eukaryoten dauerhaft an den Telomeren vor und vorübergehend unter anderem bei der Replikation, der Transkription, der Rekombination und der DNA-Reparatur vor, z. B. das Einzelstrang-bindende Protein und manche DNA-Reparaturenzyme.
Bindung doppelsträngiger DNA
Doppelsträngige DNA mit komplementärer Basenpaarung bildet eine Doppelhelix aus (B-DNA). Diese DNA-Doppelhelix besitzt eine große und eine kleine Furche. Die kleine Furche besitzt einen kleineren Anteil der Nukleinbasen in der Oberfläche des Moleküls, weshalb sie sich weniger für eine Sequenz-spezifische Bindung eignet. Verschiedene DNA-bindende Moleküle wie Lexitropsine, Netropsin,Distamycin, Hoechst 33342, Pentamidin, DAPI oder SYBR Green I binden sequenzunabhängig an die kleine Furche doppelsträngiger DNA. Doppelstrang-bindende Proteine sind unter anderem Histone bzw. DNA-bindendes Protein H-NS,High-Mobility-Group-Proteine,DNA-Polymerasen, DNA-abhängige RNA-Polymerasen, Helicasen, Topoisomerasen, Gyrasen, Ligasen, Polynukleotid-Kinasen, Nukleasen, manche DNA-Reparaturenzyme. Sequenzspezifisch dsDNA-bindende Proteine sind unter anderem Transkriptionsfaktoren und manche Endonukleasen. Prokaryotische Transkriptionsfaktoren sind meistens kleiner als die von ihnen kontrollierten Genexpressionsprodukte, während eukaryotische Transkriptionsfaktoren meistens größer als deren kontrollierten Produkte sind und gelegentlich mehrere Kopien einer DNA-bindenden Domäne besitzen.
DNA-bindende Proteindomänen
Typische Proteindomänen bei dsDNA-bindenden Proteinen sind die Zinkfingerdomäne, die AT-Haken, die DNA-Klammer und das Helix-Turn-Helix-Motiv zur DNA-Bindung oder die Leucinzipperdomäne (bZIP) zur Dimerisierung. Bei einzelsträngiger DNA wurde unter anderem die OB-Faltungsdomäne beschrieben.
Identifikation
Methoden zur Bestimmung von Protein-DNA-Wechselwirkungen (gebundene DNA-Sequenz, DNA-bindende Proteine) sind z. B. EMSA, DNase Footprinting Assay, Filterbindungstest, DPI-ELISA, DamID, SELEX, ChIP, ChIP-on-Chip oder ChIP-Seq und DAP-Seq.
Modellierung
Verschiedene Ansätze zur molekularen Modellierung wurden beschrieben. Ebenso wurden Algorithmen zur Bestimmung der gebundenen DNA-Sequenz entwickelt.
Modifikation
Im Zuge eines Proteindesigns können z. B. Zinkfingerproteine oder TALENs entworfen werden. Mit der CRISPR/Cas-Methode können anhand einer komplementären RNA-Sequenz entsprechende DNA-Sequenzen gebunden werden.
Formen
DNA (orange) mit Histonen (blau)
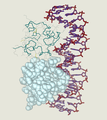
Der Lambda-Repressor mit Helix-Turn-Helix-Motiv an DNA gebunden
Das Restriktionsenzym EcoRV (grün) mit DNA.
Weblinks
- Abalone tool for modeling DNA-ligand interactions.
- DBD database of predicted transcription factors Uses a curated set of DNA-binding domains to predict transcription factors in all completely sequenced genomes
- MeSH DNA-bindende Proteine

![Der Lambda-Repressor mit Helix-Turn-Helix-Motiv an DNA gebunden[25]](http://upload.wikimedia.org/wikipedia/commons/thumb/8/8f/Lambda_repressor_1LMB.png/82px-Lambda_repressor_1LMB.png)
![Das Restriktionsenzym EcoRV (grün) mit DNA.[26]](http://upload.wikimedia.org/wikipedia/commons/thumb/d/dc/EcoRV_1RVA.png/120px-EcoRV_1RVA.png)
