Hepatitis-B-Virus
| Hepatitis-B-Virus | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|

3D-Rekonstruktion | ||||||||||||||||||
| Systematik | ||||||||||||||||||
| ||||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||||
| ||||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||||
| Hepatitis B virus | ||||||||||||||||||
| Kurzbezeichnung | ||||||||||||||||||
| HBV | ||||||||||||||||||
| Links | ||||||||||||||||||
|
Das Hepatitis-B-Virus (HBV) ist ein behülltes DNA-Virus mit überwiegend doppelsträngigem Genom und der Auslöser der Hepatitis B. Das Hepatitis-B-Virus entstammt der Gattung der Orthohepadnaviren innerhalb der Familie (Biologie) der Hepadnaviridae.
Inhaltsverzeichnis
Erkrankung
HBV vermehrt sich nahezu ausschließlich in Leberzellen. Es ist zwar selbst nicht zytopathogen und verursacht daher keinen zytopathischen Effekt; so verlieren Leberzellen nicht ihre Funktion, auch wenn sie sehr viele Viren herstellen. Jedoch verursacht die Immunreaktion eine zunehmende Schädigung der Hepatozyten. Eine zu starke Reaktion kann eine fulminante Hepatitis zur Folge haben. Zusätzlich zur Hepatitis B verursacht HBV in chronischen Verläufen (bei einer zu schwachen Immunreaktion) durch die fortlaufende Entzündung eine Leberfibrose und dann Leberzirrhose. Als Folge der Zirrhose kann Leberkrebs entstehen. Durch die kontinuierliche Wachstumsstimulation zum Ersetzen der durch das Immunsystem zerstörten infizierten Hepatozyten und durch die Zytokine mit teilweise wachstumsfördernden Eigenschaften und durch die teilweise Insertion in die zelluläre DNA entstehen vermehrt Kopierfehler in der DNA während der Mitose (in der S-Phase), was ebenfalls die Entstehung von Tumoren begünstigt.
Möglicherweise existiert auch ein Zusammenhang mit anderen Krebsarten wie Pankreaskrebs.
Aufbau
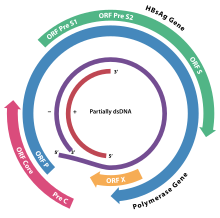
Das Virion besitzt eine Virushülle und ein 28 nm großes, ikosaedrisches Kapsid. Das Kapsid ist aus 180 Kopien des HBV-Kernproteins (auch hepatitis B core antigen HBcAg) aufgebaut und umschließt die virale DNA und eine DNA-abhängige DNA-Polymerase mit einer Reverse-Transkriptase-Aktivität (RT), ähnlich einer retroviralen Polymerase. Die Virushülle enthält als Membranprotein das HBsAg und das Pre-S1-Protein zur Bindung an die Wirtszelle. Mit einem Durchmesser von 42 nm gehört das sphärische HBV (auch Dane particle genannt) eher zu den kleineren Tierviren.
Es existieren auch nicht-infektiöse filamentöse Formen ohne Kapsid von 22 nm Durchmesser als Sphäre bzw. auch als Röhrenform, da HBsAg im Überschuss in der Leber Infizierter gebildet wird und im Blut zirkulieren. Diese Virosomen bestehen aus HBsAg in einer Lipiddoppelschicht. Aufgrund der Immunogenität ohne Infektiosität wurden sie bei der ersten Generation von Hepatitis-B-Impfstoffen als sogenannte Plasmaimpfstoffe verwendet. Mittlerweile werden HBV-Impfstoffe gentechnisch erzeugt. Das HBcAg kann bei rekombinanter Expression (ohne HBsAg) ebenfalls Virosomen bilden.
HBV erreicht im Blut eine Viruskonzentration zwischen 104 und 109 Virionen pro Milliliter, die Virosomen aus HBsAg erreichen sogar Partikelkonzentrationen bis zu 1012 Partikel pro Milliliter.
Genom
Das Genom des HBV besteht aus zirkulär-geschlossener DNA, jedoch ist die DNA nicht vollständig doppelsträngig und ein Strang durchgängig. Ein Ende des durchgängigen Strangs von circa 3020–3320 Nukleotide bindet die virale DNA-Polymerase, während der kürzere Strang mit 1700 bis 2800 Nukleotiden deutlich kürzer ist.
Der durchgängige (–)-Strang (nicht-codierend) ist revers-komplementär zur viralen mRNA. Die virale DNA ist bereits kurz nach der Infektion im Zellkern nachweisbar. Die teilweise doppelsträngige DNA wird am (+)-Strang vervollständigt und eine RNA-Sequenz am (+)-Strang entfernt. Zuletzt werden überhängende Nukleotide entfernt und der (–)-Strang zum Ring geschlossen.
Die vier Gene des HBV werden als C (core), X, P (polymerase) und S (surface) bezeichnet. Das core-Protein HBcAg wird aus einem Präprotein durch Proteolyse erzeugt. Das Gen S des Proteins HBsAg enthält drei Leseraster-konforme Start-Codons (AUG), wodurch drei Proteine entstehen, pre-S1 (synonym L), pre-S2 (synonym M) und S.
Verschiedene nicht-codierende RNA wurden im Genom des HBV identifiziert. Diese werden als Hepatitis B virus PRE alpha, Hepatitis B virus PRE beta und HBV RNA encapsidation signal epsilon bezeichnet.
Proteine
Das Virion des HBV besteht aus vier strukturellen Proteinen, weiterhin kommt das lösliche HBeAg außerhalb des Virions vor.
HBsAg
HBsAg (englisch HBV surface antigen ‚HBV-Oberflächenantigen‘) ist ein Oberflächenprotein des Virions, das im Zuge der Immunreaktion von Antikörpern als Antigen erkannt wird. Es ist durch eine virologische Diagnostik im Blut von Infizierten nachweisbar und zeigt an, ab wann ein Patient nicht mehr ansteckend ist. In der Histologie wird HBsAg durch eine Shikata-Orceinfärbung nachgewiesen.
Historisch wurde HBsAg zuerst als Australia Antigen bekannt, da es zuerst von Baruch Samuel Blumberg im Blutserum von australischen Aborigines entdeckt wurde.Alfred Prince entdeckte 1968 den Zusammenhang mit einer Hepatitisform. Der erste Impfstoff gegen HBV (Heptavax) wurde aus dem Blutplasma von HBV-Infizierten gereinigt, aufgrund des Kontaminationsrisikos durch andere Pathogene wurde später auf rekombinante Proteine aus der Bäckerhefe umgestellt.
Das Hepatitis-D-Virus benötigt HBsAg, um pathogen zu werden.
HBcAg
HBcAg (englisch core antigen ‚Antigen des Viruskerns‘) dient der Verpackung der viralen DNA und wird zum Nachweis einer momentanen Infektion verwendet. Es ist das Kapsidprotein des HBV.
HBeAg
HBeAg (englisch extracellular antigen ‚extrazelluläres Antigen‘) ist eine Spleißvariante aus den offenen Leserastern core-ORF und pre-C-ORF. HBeAg wird sezerniert und kommt nicht im Virion vor. HBeAg wird ebenso wie HBcAg zum Nachweis einer momentanen Infektion eingesetzt. Vermutlich dient es der Minderung der Genexpression von Toll-like Rezeptor 2 auf Hepatozyten und Monozyten und somit einer Dämpfung der Immunantwort gegen HBV. HBeAg ist nicht essentiell, d. h. Virionen ohne eine HBeAg-Expression sind infektiös und pathogen.
Hepatitis-B-Virus DNA-Polymerase
Die DNA-Polymerase des HBV dient der Replikation der viralen Genoms. Lamivudine und Penciclovir hemmen bei nichtresistenten HBV-Stämmen die Polymerase.
HBx
Das X-Protein HBx ist ein kleiner transkriptioneller Transaktivator, der die zelluläre Transkription von HBV-Genen reguliert. Darüber hinaus ändert er den Zellzyklus und bildet einen Proteinkomplex mit dem zellulären Protein HBX interacting protein (HBXIP), der mit dem Spindelapparat während der Zellteilung interagiert.
Möglicherweise entstammt das X-Protein einer DNA-Glykosylase. HBx erhöht die Ausschüttung von Calciumionen aus den Mitochondrien. Weiterhin interagiert HBx mit Damaged DNA Binding Protein 1 (DDB1) und hemmt die CUL4-DDB1-E3-Komplexe und dadurch das Ubiquitin-Proteasom-System. HBx fördert die Zellteilung und fördert dadurch die Entstehung von Tumoren. HBx hemmt die Protein-Arginin-Methyltransferase PRMT1, was die Replikation des HBV verstärkt.
Vorkommen und Genotypen
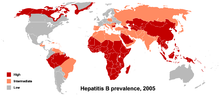
Der Reservoirwirt des HBV ist hauptsächlich der Mensch, daneben kommt HBV auch in Menschenaffen und Gibbons vor. Die mindestens neun bekannten Genotypen des HBV werden A bis I benannt. Möglicherweise existiert auch ein Genotyp J, der in auf den Ryūkyū-Inseln in Japan identifiziert wurde. Die unterschiedlichen Genotypen sprechen unterschiedlich auf Therapien an.
Die Genotypen unterscheiden sich mindestens um acht Prozent auf der Genomebene und haben unterschiedliche geographische Häufigkeiten. Der Genotyp F unterscheidet sich von den anderen am stärksten, mit vierzehn Prozent Unterschied. Der Genotyp A ist prävalent in Europa, Afrika und Südasien, einschließlich der Philippinen. Die Genotypen B und C sind vorherrschend in Asien; der Genotyp D tritt im Mittelmeerraum, dem Nahen Osten und Indien auf. Der Genotyp E existiert in Afrika südlich der Sahara. Der Genotyp F (oder H) kommt in Zentral- und Südamerika vor. Der Genotyp G wurde in Frankreich und Deutschland gefunden. Die Genotypen A, D und F dominieren in Brasilien und alle Genotypen kommen mit ethnischen Häufungen in den Vereinigten Staaten vor.
Innerhalb der Genotypen (außer E und G) sind Subtypen beschrieben, die in ihrer DNA-Sequenz untereinander um vier bis acht Prozent abweichen.
- Genotyp A hat zwei Subtypen: Aa (A1) in Afrika/Asien und Ae (A2) in Europa/USA. Letzterer tritt auch bei mittels Hefezellen gewonnenen Impfstoffen auf, der auch vor Infektionen anderer Genotypen schützt.
- Genotyp B besitzt zwei geographisch getrennte Subtypen: Bj/B1 ('j' — Japan) und Ba/B2 ('a' — Asien). Der Genotyp Ba wird weiter in vier clades unterteilt (B2 — B4).
- Genotyp C hat ebenso zwei geographisch getrennte Subtypen: Cs (C1) in Südostasien und Ce (C2) in Ostasien. Der C-Subtyp wird in fünf clades unterteilt, während ein möglicher Sechster (C6) und ein Siebter (C7) bisher nur in einem Isolat in den Philippinen und auf West-Papua gefunden wurden. Der Genotyp C1 kommt in Vietnam, Myanmar und Thailand vor. Der Genotyp C2 tritt in Japan, Korea und China auf. Der Genotyp C3 wird in Neukaledonien und Polynesien gefunden. Der Genotyp C4 kommt in Australien vor und C5 auf den Philippinen.
- Genotyp D wird in sieben Subtypen unterteilt (D1 — D7). Er ist in Afrika, Europa, im Mittelmeerraum und Indien verbreitet.
- Genotyp E hat nur einen Subtyp, er tritt in West- und Südafrika auf.
- Genotyp F (dominant in Süd- und Mittelamerika) ist in vier Subtypen unterteilt (F1 — F4). Der Subtyp F1 wird weiter in die clades 1a und 1b getrennt. In Venezuela werden die Subtypen F1, F2 und F3 im Westen und Osten gefunden, während F3 im Süden alleinig auftritt. Der Subtyp Ia kommt in Mittelamerika, der Subtyp III im nördlichen Südamerika und IV im südlichen Südamerika vor. Das clade Ib wird auf beinahe dem ganzen amerikanischen Kontinent (außer dem nördlichen Südamerika) und das clade II wird in Zentral- und Südamerika gefunden.
- Genotyp G hat nur einen Subtyp, er wurde in Frankreich und den USA beobachtet.
- Genotyp H tritt in Mexiko und Zentralamerika auf.
- Genotyp I wurde in Vietnam und Laos identifiziert.
Evolution
Die genaue Bestimmung der frühen Evolution des HBV ist problematisch. Die Aufspaltung der Hepadnaviren in Ortho- und Avihepadnaviren erfolgte vor ungefähr 125.000 Jahren (95 % Intervall 78,297–313,500). Sowohl Avi- als auch Orthohepadnaviren begannen etwa vor 25.000 Jahren, sich stärker zu verändern. Zu dieser Zeit erfolgte die Aufspaltung in die acht Genotype. Humane Hepadnaviren trennten sich etwa vor 7.000 (95 % Intervall: 5,287–9,270) bis 10.000 Jahren (95 % Intervall: 6,305–16,681). Den Avihepadnaviren fehlt das X-Protein, jedoch sind noch Reste im Entenhepadnavirus zu finden. Die Rate nicht-synonymer Mutationen des HBV wurde auf etwa 2×10−5 Aminosäuresubstitutionen pro Stelle pro Jahr geschätzt. Die mittlere Häufigkeit von Nukleotidsubstitutionen pro Stelle pro Jahr wurde mit etwa 7.9 × 10−5 beziffert. Eine andere Schätzung vermutet den letzten gemeinsamen Vorfahren der humanen HBV-Stämme vor 1.500 Jahren und eine Trennung von den Avihepadnaviren vor 6.000 Jahren bei einer Mutationsrate von etwa 10−6 Substitutionen pro Stelle pro Jahr.
Klassifizierung
HBV ist ein typischer Vertreter der Gattung der Orthohepadnaviren, die drei weitere Vertreter aufweist: das Ground squirrel hepatitis virus, das Woodchuck hepatitis virus, und das Woolly monkey hepatitis B virus. Die Familie der Hepadnaviridae enthält noch zwei weitere Gattungen, die Avihepadnaviren und eine noch zu Benennende. Die Familie ist noch keiner Ordnung zugeordnet. HBV-ähnliche Viren wurden bereits in allen Altweltaffen gefunden (Orang-Utans, Gibbons, Gorillas und Schimpansen) und auch in Neuweltaffen wie dem Wollaffen nachgewiesen, was einen älteren Ursprung dieses Virus in Primaten nahelegt.
HBV werden in vier Serotypen (adr, adw, ayr, ayw) eingeteilt, je nach den antigenen Epitopen auf ihren Hüllproteinen, genetisch werden sie, je nach ihren Mutationen, acht Genotypen (A–H) zugeordnet. Die einzelnen Genotypen haben eine unterschiedliche geografische Verteilung und werden zur epidemiologischen Bestimmung der Transmission und der viralen Evolution herangezogen. Die unterschiedlichen Genotypen weisen auch Unterschiede in den Krankheitsverläufen, der Häufigkeit von Komplikationen sowie in der Therapie- und der Impfstoffwahl auf.
Replikationszyklus
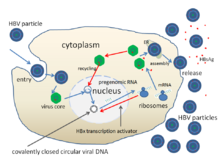
Der Replikationszyklus des HBV:
- Niedrig affine Anheftung (engl. adsorption) an die Zellmembran über Glykosaminoglykane, dann Bindung an NTCP (Na+-taurocholate cotransporting polypeptide) und anschließende Endozytose über einen bislang ungeklärten Mechanismus.
- Penetration (engl. penetration oder uncoating) durch eine induzierte Fusion der Virushülle und der Endosomenmembran, wodurch das core mit der DNA des Virus ins Zytosol freigesetzt wird.
- Entpacken (engl. uncoating) und Transport in den Zellkern über Chaperone. Dadurch erfolgt die Freisetzung der DNA aus dem core und anschließend das Vervollständigen des (–)-Strangs und der Ringschluss zur cccDNA, welche als Vorlage für die Transkription der vier viralen mRNA (Pre-S1/L, Pre-S2/M, S und X) und der beiden zur Expression und Genomsynthese verwendeten RNA (C/P und Pre-C) dient. Alle viralen RNA tragen eine Cap-Struktur und einen Poly-A-Schwanz.
- Replikation (engl. replication) im Zytosol, bei der die längste RNA (C/P, länger als das virale DNA-Genom) als Vorlage für die Synthese des viralen DNA-Genoms dient, sowie zur Proteinbiosynthese des Kapsids und der viralen Polymerase. Die RNA-Zwischenstufe erhöht die Mutationsrate zur Erzeugung von Fluchtmutationen. Die cccDNA wird bis zum Zeitpunkt der Synthese der Hüllproteine HBcAg und HBsAg (nach der Replikation) in den Zellkern importiert, erst durch die Hüllproteine erfolgt ein Export der cccDNA aus dem Zellkern. Durch den Kernimport des Genoms entsteht durch die Rückstellung unverpackter viraler Genome im Zellkern die gelegentlich auftretende persistente Infektion.
- Zusammenbau (engl. assembly) der neuen Virionen, auf den eine Abschnürung aus der Zelle oder ein Rücktransport zum Zellkern folgt, wo die DNA vom HBcAg getrennt wird und eine erneute Genomsynthese beginnt.
- Freisetzung (engl. budding, release) der Tochtervirionen durch Exozytose (nicht-lytisch) bei gleichzeitiger Synthese des DNA-Genoms im Zytoplasma aus der längsten mRNA durch die RT-Aktivität der viralen Polymerase.
Umweltstabilität
Als behülltes Virus ist das Hepatitis-B-Virus empfindlich gegenüber chemischen oder physikalischen Desinfektionsverfahren, wird jedoch aufgrund des hohen Proteinanteils in der Virushülle im Vergleich zu anderen behüllten Viren durch Alkohole oder Tenside nur relativ langsam inaktiviert. Die Inaktivierung von HBV durch Desinfektionsverfahren kann mit dem MAD-Test bestimmt werden.
Meldepflicht
In Deutschland ist jeder direkte oder indirekte Nachweis vom Hepatitis-B-Virus namentlich meldepflichtig nach § 7 des Infektionsschutzgesetzes.
In der Schweiz ist der positive laboranalytische Befund zu einem Hepatitis-B-Virus meldepflichtig und zwar nach dem Epidemiengesetz (EpG) in Verbindung mit der Epidemienverordnung und Anhang 3 der Verordnung des EDI über die Meldung von Beobachtungen übertragbarer Krankheiten des Menschen.
Literatur
- S. Modrow, Dietrich Falke, U. Truyen: Molekulare Virologie. 2. Auflage. Spektrum, Heidelberg 2003, ISBN 3-8274-1086-X.
- D. M. Knipe, Peter M. Howley (Hrsg.): Fields Virology. 5. Auflage. Philadelphia 2007, ISBN 978-0-7817-6060-7.
Weblinks
- Hepatitis B und D, RKI, 20. Juni 2016